
生命科学学院 新闻动态
北京时间2024年1月9日,西湖大学施一公团队在《自然结构与分子生物学》(Nature Structural and Molecular Biology)在线发表了题为“Structural Insights into Branch Site Proofreading by Human Spliceosome”的最新研究论文,报道了人源17S U2 snRNP复合物和剪接体pre-A复合物的高分辨率结构,并结合生化和功能实验,揭示了剪接体进行分支位点选择、校正的分子机理。
文章链接:
https://www.nature.com/articles/s41594-023-01188-0
文章截图:

mRNA剪接是真核生物基因表达的关键步骤,由剪接体催化完成。剪接体是一个巨大且高度动态的分子机器,其核心组成元件为5种核内小核糖核蛋白复合物(snRNP),分别为U1,U2,U4,U5和U6 snRNP。剪接体通过识别pre-mRNA上保守的剪接位点完成早期的组装。首先,U1 snRNP识别5'剪接位点,剪接因子SF1识别分支位点(branch site, BS),形成E complex。随后,U2 snRNP替换SF1,识别分支位点,并且U2 snRNA与分支位点形成RNA双螺旋,完成A complex的组装1。在高等真核生物中,大部分基因都会发生可变剪接,这个过程需要剪接体选择不同的剪接位点。因此,研究这些早期复合物对于理解剪接反应的保真性和调控机制至关重要。近些年,随着冷冻电镜技术的突破,一系列人源和酵母的剪接体复合物结构获得解析,极大地促进了人们对mRNA剪接分子机制的认识2-5。但由于剪接反应早期的复合物具有高度的动态性和不稳定性,其结构研究进展缓慢。
研究人员首先通过PRP5上引入的FLAG标签纯化出内源的17S U2 snRNP并解析了整体分辨率达到2.5 Å的电镜结构。随后通过使用exon-definition的pre-mRNA底物和小分子抑制剂spliceostatin A (SSA),成功捕获了一个介于E和A complex之间的中间反应状态,命名为pre-A复合物。Pre-A复合物主要包含两个区域:U2 snRNP区域和U1 snRNP区域。其中U1 snRNP非常柔性,未能解析到较高的分辨率,而U2 snRNP区域整体分辨率达到3Å,可以搭建原子模型(图1)。
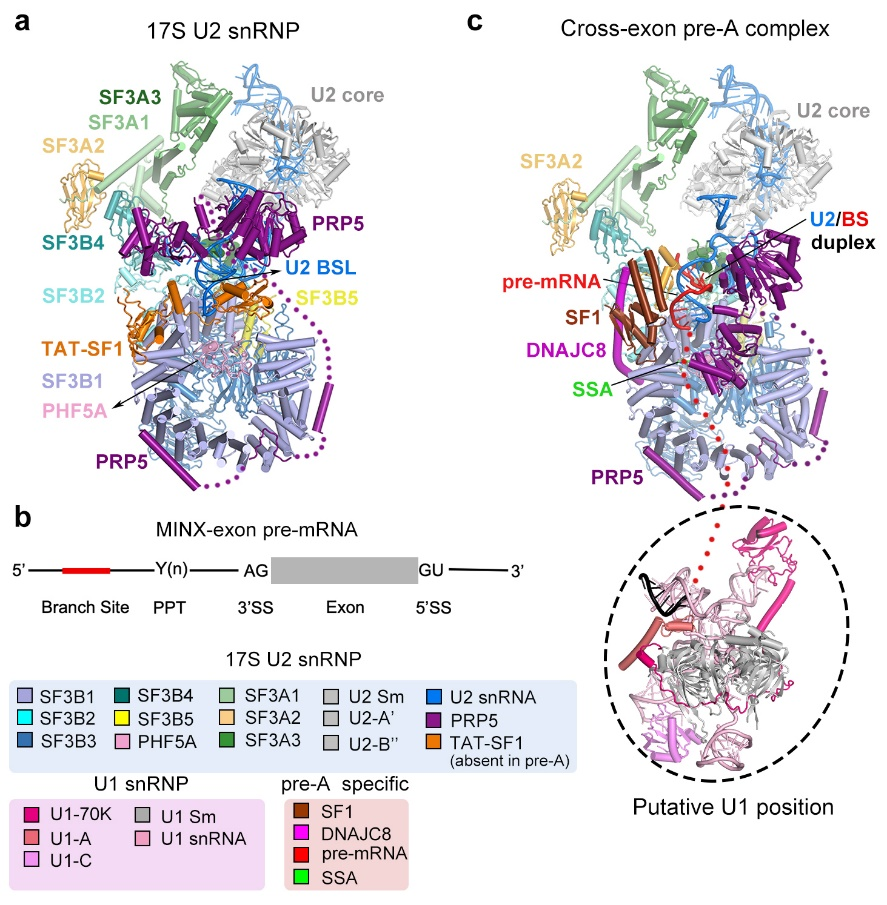
图1: 17S U2 snRNP和pre-A复合物的电镜结构
在17S U2 snRNP中PRP5主要通过其N端锚定在SF3B1外围,其中acidic loop占据了SF3B1的RNA通道,而它的解旋酶结构域则结合在U2 snRNA附近。剪接因子TAT-SF1通过其Linker domain将U2 snRNA的BS-interacting stem loop (BSL)包裹起来,从而避免BSL过早打开以及结合pre-mRNA。在pre-A复合物中,SF3B1仍然处于开放状态,PRP5的acidic loop依然结合在SF3B1的RNA通道中,但其解旋酶结构域发生了显著的构象变化。与之相应的,TAT-SF1从复合物中解离,并且U2 snRNA和pre-mRNA通过部分的互补配对,形成了一个初始的双链结构(initial U2/BS duplex)。剪接因子SF1和DNAJC8稳定了局部的构象。
同先前报道的剪接反应抑制剂E7107一样6, SSA也结合在SF3B1和PHF5A形成的铰链口袋(hinged pocket)中。不同的是,SSA与PHF5A第26位的半胱氨酸形成了共价键。由于SSA的结合,导致分支位点腺苷(BS-A)无法进入铰链口袋而游离在外面。与此同时,分支位点下游的多聚嘧啶区(polypyrimidine tract)也没有替换PRP5的acidic loop结合到SF3B1的RNA通道path中。据此,研究人员推测,BS-A进入铰链口袋是诱发SF3B1从开放到关闭构象变化的初始条件,而伴随着SF3B1的构象变化,PRP5、SF1以及DNAJC8会发生解离,SF3B1紧紧包裹住pre-mRNA,从而完成U2 snRNP对分支位点的选择,形成A complex(图2)。在这个过程中, PRP5的acidic loop与PPT竞争结合SF3B1的RNA通道是PRP5行使其校正功能的关键。
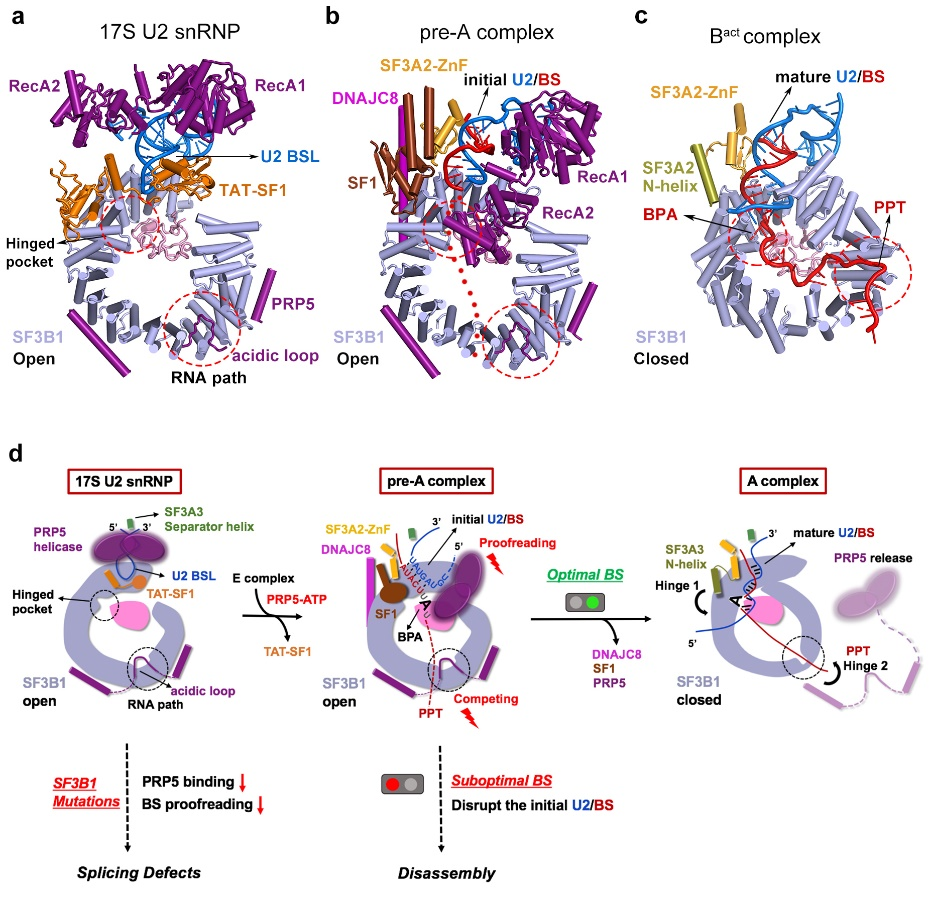
图2:
17S U2 snRNP到A complex过程中的结构变化以及PRP5校正U2/BS duplex的模型图
值得关注的是,研究人员发现PRP5的acidic loop与SF3B1的结合界面上,分布着大量与癌症相关的突变位点。结合先前对PRP5功能7和癌症患者中剪接异常现象8的研究,研究人员推测这些突变可能是通过削弱PRR5的结合,影响其对分支位点的校正功能,进而引发剪接异常,导致疾病的发生。研究人员的生化和RNA-seq数据支持了这种观点。
总的来说,本研究为理解剪接体的早期组装过程以及分支位点的识别和选择机制提供了重要的结构信息,同时也为与癌症相关的SF3B1突变的致病机制提供了新的见解。
西湖大学生命科学学院施一公教授为本文的通讯作者,原西湖大学助理研究员张晓峰(现为中国科学技术大学研究员)为本文的共同通讯作者。张晓峰、西湖大学副研究员占谢超,博士生卞彤和杨丰华为本文的共同第一作者。西湖大学博士生卢怡辰、邢芷涵、西湖大学科研助理范荣艳、Foghorn Therapeutics公司Nicolas A. Larsen参与了本研究。清华大学张强锋教授和李盼博士在RNA测序上提供了诸多帮助。电镜数据采集于西湖大学冷冻电镜平台,计算工作得到西湖大学高性能计算平台的支持,质谱分析得到西湖大学质谱与代谢组学平台的支持。本研究获得了国家自然科学基金委、西湖教育基金会、西湖大学、西湖实验室、中国博士后科学基金、国家创新型人才博士后计划的相关经费支持。
参考文献:
1. Will, C.L. & Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology 3(2011).
2. Kastner, B., Will, C.L., Stark, H. & Luhrmann, R. Structural Insights into Nuclear pre-mRNA Splicing in Higher Eukaryotes. Cold Spring Harb Perspect Biol (2019).
3. Yan, C., Wan, R. & Shi, Y. Molecular Mechanisms of pre-mRNA Splicing through Structural Biology of the Spliceosome. Cold Spring Harb Perspect Biol 11(2019).
4. Zhang, Z.W. et al. Molecular architecture of the human 17S U2 snRNP. Nature 583, 310-+ (2020).
5. Zhang, Z. et al. Structural insights into how Prp5 proofreads the pre-mRNA branch site. Nature 596, 296-300 (2021).
6. Finci, L.I. et al. The cryo-EM structure of the SF3b spliceosome complex bound to a splicing modulator reveals a pre-mRNA substrate competitive mechanism of action. Genes & development 32, 309-320 (2018).
7. Xu, Y.Z. & Query, C.C. Competition between the ATPase prp5 and branch region-U2 snRNA pairing modulates the fidelity of spliceosome assembly. Molecular Cell 28, 838-849 (2007).
8. Alsafadi, S. et al. Cancer-associated SF3B1 mutations affect alternative splicing by promoting alternative branchpoint usage. Nature Communications 7(2016).
最新资讯
















